Introduzione
Sequenze di allele stati o “motivi” sulla base di polimorfismi a il genoma mitocondriale (mtDNA) e non ricombinante porzione del cromosoma Y (NRY) sono stati proposti per tracciare ascendenze Ebraiche. In particolare, Skorecki et al., (1997) in primo luogo suggerito, che la distribuzione differenziale degli aplotipi Y-DNA basati su due marcatori, il polimorfismo Y Alu (YAP) e la Y-STR (Short Tandem Repeat) DYS19, potrebbe essere un proxy della discesa patrilineare di Cohanim sommi sacerdoti. L’aplotipo YAP-/DYS19 B è stato riconosciuto come il possibile aplotipo fondatore del sacerdozio ebraico. Poco dopo, Thomas et al. (1998) ha affinato questa ipotesi sulla base della variabilità a 6 Y-SNPs (polimorfismi a singolo nucleotide) e 6 Y-STR., Un aplotipo modale è stato trovato nei cromosomi Y di Cohen insieme a un gruppo di aplotipi strettamente correlati, che appartenessero a comunità Ashkenazite o sefardite, la cui coalescenza è stata datata all’incirca al tempo del Regno di David (2619-3221 anni fa). Nebel et al. (2000, 2001) ha definito l’aplotipo Modale di Cohen o CMH (Tabella 1) come un aplotipo 6-locus Y-STR appartenente all’aplogruppo Eu10, cioè J1-M267 secondo la nomenclatura attuale (Y-DNA Haplogroup Tree 2014 Version: 9.70)., Tale motivo ha portato a 2-3 passi mutazionali rispetto ad altri aplotipi modali Eu10 osservati in gruppi di lingua araba (MH Galilea, MH Beduini, MH palestinesi) e ad un passo dall’aplotipo modale parafiletico dei curdi musulmani appartenenti all’aplogruppo Eu9 (J2-M172 nell’attuale nomenclatura). In uno studio successivo Thomas e colleghi hanno riportato motivi ebraici CMH anche da un insieme di aplotipi del Sud-est africano (Thomas et al., 2000).
Nel loro studio più recente sulla questione, Hammer et al., (2009) hanno cercato di indagare ulteriormente la prova Y-DNA della discendenza biblica di Cohanim da un singolo antenato (il biblico Aaron) estendendo il potere di discriminazione del CMH da 6 a 12 – locus Y-STR aplotipi (eCMH, Tabella 1). Hanno affermato che l’origine della diversità associata ai cromosomi Cohanim J1-P58 potrebbe essere datata tra 4280 e 2100 anni fa.

Tabella 1. NRY motivi aplotipici.,
Gli aplotipi modali levitici (LMH e eLMH, Tabella 1) furono invece proposti da Behar et al. (2003) come aplotipi modali alternativi Y-STR all’interno della variazione dell’aplogruppo R1a-M17 dei leviti Ashkenazim, in parte condivisa con gli europei orientali. Questa scoperta ha innescato l’ipotesi dell’origine dello yiddish da una lingua slava (sorabo) e l’introgressione dei cromosomi Y khazariani nella formazione iniziale degli ebrei ashkenaziti circa 1000 anni fa., Tuttavia, le analisi di resequencing hanno trovato vari eventi fondatori tra i leviti ashkenaziti all’interno di R1a dimostrando che un particolare sotto-clade, l’M582, sarebbe una firma di origine vicino orientale in tempi pre-diaspora (Rootsi et al., 2013).
Thomas et al. (2002) ha trovato segnali di eventi fondanti molto più forti in lignaggi specifici per le donne (mtDNA) di diverse comunità ebraiche rispetto ai corrispondenti lignaggi specifici per gli uomini., Hanno trovato almeno 8 aplotipi modali (frequenza >10%) nella regione HVS-I del DNA mitocondriale in 8 comunità di ebrei geograficamente separate, mentre non è stata osservata alcuna differenziazione negli aplotipi Y-DNA delle popolazioni ebree e ospiti.
Utilizzando un’analisi ad alta risoluzione degli aplogruppi K e N, Behar e i suoi collaboratori identificarono quattro lignaggi fondatori del mtDNA come la fonte matrilineare di circa il 40% dell’attuale popolazione di ebrei ashkenaziti (Behar et al., 2006)., Tali lignaggi sono stati descritti come originari del Medio Oriente circa 2100 anni fa ” probabilmente da una piscina mtDNA ebraica / levantina.”Tuttavia, la complessa origine matrilineare degli Ashkenazi sembra essere stata meglio rappresentata da Costa et al. (2013), che ha rivelato che la grande maggioranza dei lignaggi materni ashkenaziti erano il risultato di eventi reiterati di mescolanza all’interno dell’Europa. Per quanto riguarda i potenziali confonditori demografici dello scenario di cui sopra, Behar et al. (2004) e Guha et al., (2012) hanno affermato una forte deriva genetica per contribuire alla frequenza insolitamente elevata di alleli recessivi e bassa diversità di mtDNA e Y-DNA nelle popolazioni ashkenazite. D’altra parte, la recente letteratura basata su analisi genome-wide (Atzmon et al., 2010; Behar et al., 2010; Bray et al., 2010) ha evidenziato il ruolo decisivo della mescolanza nel plasmare l’attuale pool di DNA ebraico.
Negli ultimi anni le aziende genealogiche genetiche hanno reclutato decine di migliaia di volontari che hanno accettato di condividere i risultati dei loro test genetici in archivi pubblici., Il più grande database ancestry disponibile fino ad oggi è il Family Tree DNA archive (FtDNA), che include più di 506 K record per Y-DNA e circa 180 K record per mtDNA suddivisi in quasi 8 K progetti, in cui i partecipanti possono condividere il proprio profilo del DNA per tracciare un patrimonio comune per cognome, lignaggio o geografia. Facendo uso di tale risorsa preziosa, abbiamo costruito un ampio database di profili mtDNA e Y-DNA disponibili da >600 progetti FtDNA., Abbiamo esplorato il database per esaminare la variabilità associata ai motivi genetici proposti dalla letteratura in volontari che rivendicano una discendenza ebraica e in gruppi non ebrei di origine europea, africana o mediorientale.
Abbiamo cercato di fornire un background sperimentale aggiornato con cui argomentare i difetti e le insidie che si possono incontrare quando si usano motivi aplotipici come marcatori diagnostici di ascendenza ebraica.,
Materiali e metodi
Creazione di database
Gli aplotipi Y-STR e Y-SNP sono stati scaricati dal browser Family Tree DNA Y Chromosome (https://www.familytreedna.com/projects.aspx). Per abbinare motivi di riferimento (Behar et al., 2003) con standard nomenclature guidelines (Gusmão et al., 2006) gli stati allelici a DYS439 sono stati corretti sottraendo 4 ripetizioni. Gli aplotipi HVS-I e gli SNP mtDNA sono stati scaricati dal Family Tree DNA mt Chromosome Browser (https://www.familytreedna.com/projects.aspx).,
I record (https://www.familytreedna.com/projects.aspx) furono inizialmente divisi in Progetti volti a ricostruire esplicitamente la discendenza ebraica e altri Progetti. In secondo luogo, abbiamo filtrato all’interno e tra i Progetti quei record con numeri di kit duplicati e origini incerte. Quando non disponibile, le associazioni tra aplotipi e SNP sono state fatte per numero di kit., Infine, abbiamo rimosso gli aplotipi che non si adattavano a un doppio criterio: NRY-aplotipi-da digitare con un minimo di 12 loci Y-STR (l’insieme di motivi” estesi”) e assegnati agli aplogruppi digitando direttamente SNP o su previsioni basate su profili STR ≥25-locus; mtDNA-aplotipi-da sequenziare almeno per i siti HVS-I 16,024-16,569 e assegnati agli aplogruppi digitando direttamente. Abbiamo ottenuto una griglia finale di 62.920 record Y-DNA e 30.469 record mtDNA. Le banche dati sono state ricercate per 6 e 12 motivi Y-STR (ebraico e arabo) e per 4 motivi ashkenaziti HVS-I (Tabelle 1, 2).,

Tabella 2. Motivi aplotipici mtDNA.
Una limitazione degli archivi genealogici genetici disponibili al pubblico è l’autoassegnazione dei partecipanti a categorie sociali / etniche, come nel caso della discendenza ebraica. Un altro è la presunta parentela tra i partecipanti all’interno dello stesso gruppo etnico., Mentre il primo limite è difficile da accertare e le fonti di errore come adozioni e paternità illegittime non possono essere escluse, da un’analisi preliminare la stretta parentela sembra influenzare solo marginalmente le statistiche sommarie presentate in questo articolo rispetto a quelle dei precedenti rapporti scientifici. I record che condividevano un antenato comune erano 12 su 3903 noti (0,3%) in Progetti di ascendenza ebraica e 606 su 48.006 noti (1,5%) negli altri Progetti.,
Calcolo dell’Aplotipo Tassi di Mutazione
Abbiamo impiegato il seguente pedigree basato su tariffe:
Media tassi di mutazione per Y-aplotipo sono stati ottenuti secondo il “genealogica” metodo (Klyosov, 2009a; Rozhanskii e Klyosov, 2011) dopo la calibrazione, per tornare mutazioni in esso descritto, e di “germinale” stime unisce padre e figlio di coppie di dati dalla letteratura (29 studi rivisti da Burgarella e Navascue s, 2011, più di Ballantyne et al., 2010).,
Nel primo caso la pendenza del diagramma di calibrazione per i 12-locus STRs dei motivi eCMH e eLMH si adatta bene ad un valore della costante del tasso di mutazione di 0,00166 mutazione per marcatore per generazione condizionale di 25 anni, cioè 0,020 mutazioni per aplotipo ogni 25 anni.
In quest’ultimo caso il numero di mutazioni osservate in un totale di 126.873 meioses (Tabella S1) ha dato un tasso medio di 2,113 × 10-3 (± 1,369 × 10-3) mutazioni per marcatore per generazione, cioè 0,025 ± 0,016 mutazioni per aplotipo per generazione., Per il locus trimerico DYS426, in cui non sono state osservate mutazioni tra i pedigree, è stato utilizzato un tasso di regressione basato su un modello di popolazione logistica (0.458 × 10-3 mut/gen, Burgarella e Navascue, 2011).
Come tassi medi di mutazione per HVSI-aplotipo abbiamo adottato il tasso basato sul pedigree in Howell et al. (2003) sulla base di 11 studi dalla letteratura: 1.06 × 10-2 mut/gen assumendo un intervallo di generazione di 25 anni. È circa sette volte superiore al tasso calibrato fossile per il segmento 16,051-16,400 seguente Soares et al. (2009): 1.,42 × 10-3 mut / gen assumendo un intervallo di generazione di 25 anni.
Analisi di rete
L’uso delle reti aveva un duplice scopo: molti dei passaggi mutazionali intermedi tra aplotipi, forse oscurati da mutazioni ricorrenti o da campionamenti incompleti, potrebbero essere stati recuperati e considerati nelle stime temporali; la posizione dei motivi aplotipici sull’albero migliore ha dato un indizio della loro filogenesi.,
Le relazioni mutazionali tra i motivi Y-DNA 12-locus e i motivi HVS-I (16.024–16.569 bp) di individui di discendenza ebraica auto-riportata sono stati visualizzati mediante l’algoritmo di rete di giunzione mediana implementato nel software Network 4.612 (http://www.fluxus-engineering.com) secondo Zalloua et al. (2008). Durante la costruzione di reti, il valore predefinito (10) è stato dato a ciascun sito HVS-I e un punteggio calcolato sulla varianza stimata in ciascun locus è stato adottato per i dati Y-STR (punteggio per un determinato locus = 10 * varianza totale su tutti i loci/varianza in quel dato locus).,
Per Y-DNA, abbiamo selezionato solo aplotipi (N = 142) assegnati al lignaggio R1a1a1-M417 e le sue sottocladi a valle contenenti il motivo LMH (16-12-25-10-11-13, Tabella 1) e aplotipi (N = 73) assegnati al lignaggio J1a2b-P58 e le sue sottocladi a valle contenenti il motivo CMH (14-16-23-10-11-12, Tabella 1). Il locus DYS385 non è stato utilizzato perché gli alleli non possono essere assegnati correttamente alla regione duplicata specifica e DYS389II è stato trattato come DYS389b = DYS389II – DYS389I., I tempi di coalescenza sono stati stimati dalle reti dalla statistica di rho utilizzando” pedigree “- così come i tassi” genealogicamente ” – based.
Per mtDNA, abbiamo selezionato solo aplotipi contenenti i tre motivi K (16093C 16224C 16311C 16519C 16524G, motif_K1; 16224C 16234T 16311C 16519C, motif_K2; 16224C 16311C 16519C, motif_K3; Tabella 2). I tempi di coalescenza sono stati stimati dalle reti dalla statistica di rho utilizzando tariffe basate su” pedigree”.,
Calcolo della probabilità condizionale bayesiana di assegnazione
Il teorema di Bayes è stato usato per calcolare la probabilità condizionale di un individuo di appartenere alla popolazione ebraica dato che tali individui portano un motivo NRY o mtDNA “diagnostico”. Tale probabilità è funzione della frequenza di un dato aplotipo in ebrei e non ebrei (ottenuto dalle tabelle 3-5) e della percentuale di individui ebrei (stimata nell’ordine di 13 milioni) nella popolazione globale (7,2 miliardi di persone).

Tabella 3., Distribuzione di NRY (digitato e previsto su Y-STRs) e motivi mtDNA nel database pubblico FTDNA.
Risultati
L’analisi dei record FtDNA (Tabelle 3-5) ha confermato che nessun motivo genetico trasmesso lungo la linea materna o paterna è esclusivo degli ebrei. Tuttavia, non è stato possibile estendere questa conclusione a sottogruppi ebraici come Leviti e Cohanim perché questo stato è raramente auto-riportato nelle voci FtDNA., L’unica eccezione è il cromosoma Y “Cohen Zadokites Project” che unisce i discendenti putativi del nipote di Aaron Zadok. Lì, infatti, potremmo verificare (Tabella S2) che i partecipanti appartenessero a un totale di 6 diversi aplogruppi con J2, non J1, come modale (63%). Nessun aplotipo 12-locus è stato osservato come privato per i membri del progetto e il J1-eCMH ha riassunto solo 4 dei 59 record con origine nota (~8%).,
L’analisi dell’aplotipo Y distribuzioni in genitoriale i rami di un albero, ha confermato che, anche se c’è una netta separazione tra la distribuzione di CMH, eCMH, LMH, e eLMH tra Ebrei e non-Ebrei popolazioni (chi quadrato risultati in Tabella 4), nessun motivo è diagnostico di monofiletico aplogruppi quando la probabilità condizionale di assegnazione è stato stimato utilizzando la formula di Bayes (Figura 1). Anche i motivi Y-DNA più risolti (eLMH ed eCMH) sono stati trovati in due o più aplogruppi indipendenti i cui tempi superiori di divergenza sono stimati non meno di 30 K anni fa., Ciò potrebbe essere spiegato da un’origine così profonda e dalla successiva evoluzione senza alcun cambiamento, dagli effetti collaterali della paternità non riconosciuta, dalla conversione genica o, molto probabilmente, dal tasso di cambiamento dell’aplotipo (si prevede che circa 24 mutazioni si verifichino in 30 K anni con aplotipi Y-STR estesi) e dall’alta frequenza del polifiletismo, dall’omoplasia.

Tabella 4. Distribuzione dei motivi negli aplogruppi Y-DNA (Hg) tra 5281 Ebrei (J) e 57.639 non ebrei (NJ).,

Figura 1. Stima della potenza diagnostica di un insieme di aplotipi modali per assegnare un dato campione alla popolazione ebraica. Ogni riga mostra la probabilità condizionale (formula di Bayes) di assegnare un determinato individuo che trasporta mtDNA (A) o Ychr (B) aplotipi modali alla popolazione ebraica in funzione della proporzione di individui ebrei nel campione. I punti rossi mostrano la probabilità posteriore ottenuta quando si immette la frazione ebraica stimata della popolazione mondiale.,
vale la pena notare che le posizioni lungo gli alberi di eLMH (centrale, Figura 2A) e eCMH (periferico, Figura 2B) suggeriscono che quest’ultimo potrebbe non essere stata presente nel pool iniziale di fondatori, ma semplicemente essere il risultato di una espansione più recente.

Figura 2. Uno dei tre alberi più parsimoniosi costruiti dall’algoritmo di rete di giunzione mediana., Abbiamo usato aplotipi 12-locus Y-STR contenenti il motivo LMH assegnato al lignaggio R1a-M417 (A) e il motivo CMH assegnato al lignaggio J1-P58 (B) insieme alle loro sottocladi a valle in individui che rivendicano una discendenza ebraica. I cerchi rappresentano aplotipi, con aree proporzionali al numero di individui che contengono.
L’analisi delle distribuzioni aplotipiche mitocondriali negli ebrei indica che, almeno con l’attuale livello di risoluzione SNP, solo un motivo (K3) su quattro è stato trovato in diversi aplogruppi (Tabella 5).,
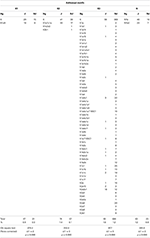
Tabella 5. Distribuzione dei quattro motivi ashkenaziti mtDNA in aplogruppi (Hg) tra 2818 Ebrei (J) e 27.651 non ebrei (NJ).
D’altra parte, i risultati di cui sopra hanno sottolineato una sovrarappresentazione di questi motivi negli ebrei rispetto ai non ebrei, così come un significativo accumulo di motivi all’interno di alcuni aplogruppi (Tabelle 4, 5)., Gli aplotipi ebraici R1a che portano il motivo levitico ashkenazita LMH (Figura 2A) sembrano condividere la variante CTS6, la cui TMRCA è stata stimata tra 1175 ± 341 anni fa (usando il tasso genealogico) e 924 ± 268 anni fa (usando il tasso della linea germinale). Inoltre, gli aplotipi ebraici J1 che mostrano il motivo Cohanim CMH (Figura 2B) sembrano condividere la variante YSC0000234, la cui TMRCA è stata stimata tra 1255 ± 441 anni fa (usando il tasso genealogico) e 986 ± 346 anni fa (usando il tasso della linea germinale)., Gli aplotipi ebraici che portano i motivi K1, K2 e N sono esclusivi, rispettivamente, degli aplogruppi K1a9, K1a1b1a e N1b (Tabella 2). I tempi di divergenza calcolati sulla variabilità osservata in questi aplogruppi più gli aplogruppi K2a2a1 hanno dato date recenti per l’antenato comune di tutti i motivi mtDNA, con quelli per K2 e K3, rispettivamente, 1370 ± 1241 anni fa e 1265 ± 639 anni fa, comparabili con i tempi dedotti per gli antenati comuni degli aplotipi Y estesi.,
Discussione
Motivi genetici costituiti da insiemi di marcatori aploidi non ricombinanti sono stati a lungo utilizzati per rintracciare le presunte origini ebraiche di singoli individui o intere popolazioni. Tali motivi uni-parentali sono stati anche proposti per assegnare ascendenza in studi di associazione e casi forensi.
Il fallimento di questa pratica è inerente alla natura della variazione genetica. Come stima conservativa possiamo aspettarci una nuova mutazione ogni 94 generazioni (28 sostituzioni ogni 2633 trasmissioni, Howell et al.,, 2003) nei lignaggi mitocondriali e circa ogni tre generazioni (3 × 10-8 mut / sito / gen in pedigree di radicazione profonda oltre 10,15 Mb, Xue et al., 2009) nei lignaggi NRY.
Pertanto, in tali sistemi genetici maggiore è il numero di marcatori tipizzati, minore è la probabilità di scoprire insiemi di alleli che corrispondono esattamente alla sequenza degli antenati fondatori., Più specificamente, una mutazione si verificherebbe ogni 40-50 trasmissioni su aplotipi Y-STR estesi, ogni 80-100 trasmissioni su aplotipi Y-STR classici a 6 locus e ogni 140-150 trasmissioni sul segmento 16.024-16.569 di aplotipi HVS–I. Quindi, dopo poche migliaia di anni potrebbero essere tutti mutati più di una volta.
D’altra parte, minore è il numero di marcatori tipizzati maggiore è la probabilità di aplotipi identici come risultato di omoplasia o di un’antica discendenza comune., In altre parole, l’adozione di aplotipi modali come marcatori per determinati eventi storici deve tenere conto della risoluzione temporale offerta dal numero di varianti incluse nelle analisi. Gli aplotipi Y definiti da decine di varianti minimizzano il rischio di identità per stato (IBS) o convergenza, consentendo quindi un elevato potere diagnostico a spese di una risoluzione temporale superficiale (TMRCA recente). Gli aplotipi definiti da 6 o 12 STR, come CMH e LMH o eCMH e eLMH, consentono una risoluzione temporale più profonda, ma con un errore maggiore dovuto a mutazioni ricorrenti., L’adozione di intere sequenze di mtDNA potrebbe migliorare la risoluzione di alcune filogenie materne. Secondo i loci many-Y-STR, tuttavia, ciò influenzerebbe necessariamente il trade-off tra il potere di discriminare tra IBD e IBS e la risoluzione temporale.
I nostri risultati sono una dimostrazione di questo argomento: l’uso di motivi aplotipici non ricombinanti come marcatori diagnostici di ascendenza ebraica si è dimostrato fortemente fuorviante quando non supportato dalla conoscenza in regioni più informative o intere sequenze., Dei motivi precedentemente assunti per tracciare antenati ebraici specifici, nessuno risultava identico per discendenza (IBD), che è ereditato senza modifiche da un antenato comune. Con poche eccezioni, motivi, sia dal NRY o il mtDNA, sono stati osservati in almeno due lignaggi indipendenti, a volte appartenenti a gruppi etnici con diverse affiliazioni culturali o geografiche., Per spiegare il modello polifiletico della distribuzione dell’aplotipo attraverso i gruppi analizzati, prevediamo, come la spiegazione più parsimoniosa, molteplici eventi fondatori e / o rimescolamento dei pool genomici attraverso la lunga storia di dispersione e mescolanza delle comunità ebraiche dalla loro fondazione.
Per fare un esempio, la firma “CMH”, nella sua versione classica ed estesa, è stata osservata in molti aplotipi di abitanti della penisola arabica con nomi tipici arabi, così come in molti ebrei appartenenti agli aplogruppi J1 e J2., La distribuzione di CMHS da etnie e aplogruppi è tale da suggerire che le conversioni geniche, adozioni e paternità illegittime potrebbero influenzare solo marginalmente i risultati a meno che non fossero molteplici e si sono verificati principalmente centinaia di anni fa.
Una spiegazione più semplice è che, tra 7600 e 10.400 anni bp (95% CI), l ‘ “Aplotipo modale di Cohen” era un aplotipo ancestrale per gli abitanti storici della penisola arabica., Circa 4000 ± 520 anni fa la popolazione ebraica stabiliva questo “aplotipo modale” insieme ai futuri arabi, che a quel tempo avevano un antenato comune con i futuri ebrei (Klyosov, 2010). Intorno al decimo secolo DC, un “CMH recente” leggermente modificato si divise dal” CMH più vecchio “(in formati aplotipici più estesi), mentre entrambi contenevano la firma 6 del” CMH”, che è ancora presente in molti aplotipi arabi. Questo “CMH recente” divenne l’aplotipo ancestrale per un lignaggio ebraico separato anche se recente all’interno dell’aplogruppo J1., Se si considerano solo gli aplotipi ” CMH ” all’interno di questa popolazione, è possibile identificare un antenato comune vissuto intorno a 1255-986 anni fa.
Concentrandosi sui motivi mitocondriali, la presenza di varianti in siti di mutazione rapida come 16.311 e 16.519 aumenta il potenziale per l’insorgenza di mutazioni ricorrenti nei segmenti HVS-I. Ciò è particolarmente critico all’interno dell’aplogruppo K., Monofilia e TMRCA recenti, vale a dire genealogie rintracciabili fino a un singolo antenato recente, potrebbe essere invocato per K1, K2, e motivi N mentre mescolanza e fondatori multipli dovrebbero essere invocati per il motivo K3 in assenza di una migliore risoluzione, in linea con genoma completo e ri-sequenziamento dei dati.
Un ulteriore elemento di incertezza è giocato dalla scelta del tasso mutazionale., L’aplotipo muta cambiando i loro alleli in modo imprevedibile, e solo un numero medio di mutazioni in un dato tempo può essere previsto con una certa probabilità, in base alle costanti del tasso di mutazione e a quanto “vecchio” è il gruppo di aplotipi in termini di un periodo dal loro antenato comune.
La questione di cui è la costante di tasso di mutazione aplotipo più adatto da applicare per tracciare percorsi storici delle popolazioni umane è stato oggetto di accesi dibattiti (vedi Soares et al., 2009; Wei et al., 2013) e la recente disponibilità di dati di intero genoma e resequencing non l’ha risolto., È ampiamente accettato che i conteggi di mutazione tra aplotipi divergenti si saturino abbastanza rapidamente a causa di mutazioni ricorrenti, specialmente nei marcatori STR. È anche implicito che i tassi calibrati su infiniti modelli di ramificazione e tempi evolutivi gonfiano TMRCA di aplotipi che sono venuti a divergere in tempi storici. È il caso del tasso proposto da Zhivotovsky et al. (2004), che è stato usato da Hammer e colleghi per sostenere che l’età di eCMHs è compatibile con la fondazione del sacerdozio Cohanim (vedi critica in Klyosov, 2009b)., Inoltre, è evidente che le stime basate sulla linea germinale, dai padri ai figli o sui pedigree profondi sono spesso supportate da statistiche molto scarse, le mutazioni osservate in alcuni marcatori sono molto poche o del tutto assenti.
A complicare l’immagine è il concetto che un tasso costante non esiste nel mondo reale. Di per sé, il numero di anni di generazione è un valore fluttuante, dipende dalle abitudini culturali, dalle opinioni religiose, dall’età alla nascita dei bambini, dalla nutrizione, dalla salute e da altre condizioni di vita su un dato territorio in un dato momento., Inoltre, la probabilità che una nuova mutazione appaia dipende dalla struttura della regione genomica in cui accade e il suo destino dipende in gran parte dalle dimensioni e dalla demografia della comunità a cui appartiene.
Di norma, il tempo più lungo per un antenato comune di un gruppo di aplotipi, meno “diagnostica” un motivo e stime temporali più incerte., Solo i gruppi con recenti antenati comuni hanno motivi piuttosto prevedibili, come è il caso degli ebrei Ashkenaziti in alcuni aplogruppi mtDNA e Y-R1a sub-cladi ampliato negli ultimi mille anni, perché non molto tempo lasciato dal antenato comune, e il suo aplotipo è ancora in giro con relativamente poche mutazioni.
Tutti i motivi proposti sono risultati essere distribuiti in modo non uniforme tra individui raggruppati in base alla loro discendenza ebraica e non ebraica, quasi sempre con un significativo arricchimento negli ebrei (Tabelle 4, 5)., Tuttavia, la composizione eterogenea degli aplotipi che li contengono ha influenzato qualsiasi tentativo di datare correttamente la loro origine. Una maggiore risoluzione di digitazione SNP e, si spera, la disponibilità di sequenze complete, potrebbe aiutare a raffinare la filogenesi di tali marcatori, in ultima analisi, chiarire il loro ruolo e il tempo dalla fondazione dei gruppi ebraici.,
In conclusione, mentre la distribuzione osservata dei sub-cladi di aplotipi presso mitocondriale e del cromosoma Y non ricombinante genomi potrebbe essere compatibile con il fondatore di eventi, in tempi recenti, all’origine di gruppi di Ebrei come Cohenite, Levita, Ashkenazite, il generale di sostanziale polyphyletism così come la loro sistematica ricorrenza in non-Ebrei gruppi mette in evidenza la mancanza di supporto per il loro utilizzo come marcatori di ascendenza Ebraica o racconti Biblici.,
Dichiarazione sul conflitto di interessi
Gli autori dichiarano che la ricerca è stata condotta in assenza di relazioni commerciali o finanziarie che potrebbero essere interpretate come un potenziale conflitto di interessi.
Riconoscimento
Gli autori vorrebbero riconoscere i revisori per i loro commenti critici e stimolanti.