Introducción
Secuencias de alelo estados o «motivos» basado en los polimorfismos en el genoma mitocondrial (adnmt) y la no-recombinación de la porción del cromosoma Y (NRY) se han propuesto para rastrear la ascendencia Judía. En particular, Skorecki et al., (1997) sugirió por primera vez, que la distribución diferencial de los haplotipos de ADN-Y basados en dos marcadores, El polimorfismo de Alu-y (Yap) y la DIS19 Y-STR (repetición corta en tándem), podría ser un proxy del descenso patrilineal de los sumos sacerdotes Cohanim. El haplotipo YAP – / DYS19 B fue reconocido como el posible haplotipo fundador del sacerdocio judío. Poco después, Thomas et al. (1998) refinaron esta hipótesis sobre la base de la variabilidad en 6 y-SNPs (polimorfismos de nucleótido único) y 6 y-STRs., Se encontró un haplotipo modal en los cromosomas de Cohen y junto con un grupo de haplotipos estrechamente relacionados si pertenecían a comunidades ashkenazíes o sefardíes, cuya coalescencia se fechó aproximadamente en la época del Reino de David (2619-3221 años atrás). Nebel et al. (2000, 2001) definieron el haplotipo modal de Cohen o CMH (Tabla 1) como un haplotipo 6-locus y-STR perteneciente al haplogrupo Eu10, que es J1-M267 según la nomenclatura actual (y-DNA haplogrupo Tree 2014 Version: 9.70)., Tal motivo resultó en 2-3 pasos mutacionales lejos de otros Haplotipos modales eu10 observados en grupos de habla árabe (MH Galilee, MH beduinos, MH palestinos) y un paso lejos del haplotipo modal parafilético de los kurdos musulmanes pertenecientes al haplogrupo Eu9 (J2-M172 en la nomenclatura actual). En un estudio posterior, Thomas y sus colegas informaron motivos judíos DE CMH también de un conjunto de haplotipos del Sudeste africano (Thomas et al., 2000).
en su estudio más reciente sobre el tema, Hammer et al., (2009) han tratado de investigar más a fondo la evidencia de ADN-Y de la descendencia Bíblica de Cohanim de un solo ancestro (el Aarón bíblico) extendiendo el poder de discriminación de la CMH de 6 a 12 locus y – STR haplotipos (eCMH, Tabla 1). Afirmaron que el origen de la diversidad asociada a los cromosomas Cohanim J1-P58 podría fecharse entre 4280 y 2100 años atrás.

la Tabla 1. NRY haplotipo motivos.,
Los Haplotipos modales Levitas (LMH y eLMH, Tabla 1) fueron propuestos por Behar et al. (2003) como haplotipos modales alternativos y-STR dentro de la variación del haplogrupo R1a-M17 de Levitas Ashkenazim, parcialmente compartidos con los europeos del Este. Este hallazgo desencadenó la hipótesis del origen del Yiddish de una lengua eslava (sorabo) y la introgresión de los cromosomas y jázaros en la formación inicial de los judíos asquenazíes hace unos 1000 años., Sin embargo, los análisis de resecuenciación encontraron varios eventos fundadores entre los levitas Ashkenazi dentro de R1a demostrando que un subclado particular, el M582, sería una firma de un origen del Cercano Oriente en tiempos previos a la Diáspora (Rootsi et al., 2013).
Thomas et al. (2002) encontraron señales de eventos fundacionales mucho más fuertes en linajes específicos para mujeres (ADNmt) de diferentes comunidades judías que en linajes específicos para hombres correspondientes., Encontraron al menos 8 haplotipos modales (frecuencia >10%) en la región HVS-I del ADN mitocondrial en 8 comunidades geográficamente separadas de judíos, mientras que no se observó diferenciación en los haplotipos de ADN-y de poblaciones judías y hospederas.
Usando un análisis de alta resolución de los haplogrupos K y N, Behar y sus colaboradores identificaron cuatro linajes fundadores del ADNmt como la fuente matrilineal de alrededor del 40% de la población actual de judíos asquenazíes (Behar et al., 2006)., Tales linajes fueron descritos como originarios de Oriente Medio hace alrededor de 2100 años » probablemente de un estanque de ADNmt hebreo / levantino.»Sin embargo, el complejo origen matrilineal de los Ashkenazi parece haber sido mejor representado por Costa et al. (2013), que reveló que la gran mayoría de los linajes maternos Ashkenazi eran el resultado de eventos de mezcla reiterada dentro de Europa. En cuanto a los posibles factores de confusión demográfica del escenario anterior, Behar et al. (2004) y Guha et al., (2012) han afirmado una fuerte deriva genética para contribuir a la inusualmente alta frecuencia de alelos de enfermedad recesiva y baja diversidad de ADNmt y ADN-Y en poblaciones Ashkenazi. Por otro lado, la literatura reciente basada en análisis de todo el genoma (Atzmon et al., 2010; Behar et al., 2010; Bray et al., 2010) destacó el papel decisivo de la mezcla en la conformación de la actual reserva de ADN judío.
en los últimos años, las compañías de genealogía genética han estado reclutando a decenas de miles de voluntarios que aceptaron compartir los resultados de sus pruebas genéticas en repositorios públicos., La base de datos de ancestros más grande disponible hasta la fecha es el Family Tree DNA archive (FtDNA), que incluye más de 506 registros K para ADN-Y y alrededor de 180 registros K para ADN-MT divididos en casi 8 proyectos K, donde los participantes pueden compartir su propio perfil de ADN para rastrear una herencia común por apellido, linaje o geografía. Haciendo uso de este valioso recurso, construimos una gran base de datos de mtDNA y perfiles de ADN-y disponibles en >600 proyectos de FtDNA., Exploramos la base de datos para estudiar la variabilidad asociada a los motivos genéticos propuestos por la literatura en voluntarios que afirman tener ascendencia judía y en grupos no judíos de origen europeo, africano o de Oriente Medio.
nuestro objetivo fue proporcionar un fondo experimental actualizado para argumentar fallas y trampas que uno puede encontrar al usar motivos de haplotipos como marcadores diagnósticos de ascendencia judía.,
materiales y métodos
construcción de bases de datos
Los haplotipos Y-STR y-SNPs fueron descargados del Explorador de cromosomas y de ADN del árbol genealógico (https://www.familytreedna.com/projects.aspx). Para que coincida con los motivos de referencia (Behar et al., 2003) con directrices de nomenclatura estándar (Gusmão et al., 2006) los Estados alélicos en DYS439 fueron corregidos restando 4 repeticiones. Los Haplotipos HVS-I y los SNP del ADNmt fueron descargados del Explorador de cromosomas mt del árbol genealógico (https://www.familytreedna.com/projects.aspx).,
Los registros (https://www.familytreedna.com/projects.aspx) se dividieron primero en proyectos destinados a reconstruir explícitamente la ascendencia judía y otros proyectos. En segundo lugar, filtramos Dentro y entre los proyectos los registros con números de kit duplicados y orígenes inciertos. Cuando no estaba disponible, las asociaciones entre haplotipos y SNPs se hicieron por número de kit., Por último, eliminamos los haplotipos que no se ajustaban a un doble criterio: NRY-haplotipos – para ser tipeados en un mínimo de 12 loci Y-STR (el conjunto de motivos «extendidos») y asignados a haplogrupos por tipificación directa SNP o sobre predicciones basadas en perfiles ≥25-locus STR; mtDNA-haplotipos—para ser secuenciados al menos para los sitios HVS-i 16,024–16,569 y asignados a haplogrupos por tipificación directa de SNPs de diagnóstico. Obtuvimos una cuadrícula final de 62,920 registros de ADN-Y y 30,469 registros de ADNmt. Se realizaron búsquedas en las bases de datos para los motivos 6 y 12 Y-STR (judíos y árabes) y para los motivos 4 HVS-I Ashkenazi (Tablas 1, 2).,

la Tabla 2. mtDNA haplotype motifs.
una limitación de los archivos genealógicos genéticos disponibles públicamente es la Auto-asignación de los participantes a categorías sociales/étnicas, como es el caso de la ascendencia judía. Otra es la supuesta relación entre los participantes dentro del mismo grupo étnico., Si bien el primer límite es difícil de determinar, y las fuentes de error como las adopciones y las paternidades ilegítimas no pueden excluirse, un análisis preliminar muestra que la estrecha relación solo afecta marginalmente a las estadísticas resumidas presentadas en este trabajo con respecto a las de informes científicos anteriores. Los registros que comparten un ancestro común fueron 12 de 3903 conocidos (0.3%) en proyectos de ascendencia judía y 606 de 48,006 conocidos (1.5%) en los otros proyectos.,
cálculo de las tasas de mutación del haplotipo
se emplearon las siguientes tasas basadas en pedigrí:
Las tasas promedio de mutación por y-haplotipo se obtuvieron de acuerdo con el método «genealógico» (Klyosov, 2009a; Rozhanskii y Klyosov, 2011) después de la calibración para las mutaciones dorsales descritas en el mismo, y a las estimaciones de «línea germinal» que combinan datos de pares de padre a hijo de la literatura (29 estudios revisados por Burgarella y navascue’S, 2011, Más Ballantyne et al., 2010).,
en el primer caso, la pendiente de la gráfica de calibración para los STRs de 12 locus de motivos eCMH y elmh se ajusta bien a un valor de la constante de tasa de mutación de 0.00166 mutaciones por marcador por generación condicional de 25 años, es decir, 0.020 mutaciones por haplotipo cada 25 años.
en este último caso, el número de mutaciones observadas en un total de 126.873 meiosis (tabla S1) dio una tasa media de 2,113 × 10-3 (± 1,369 × 10-3) mutaciones por marcador por generación, es decir, 0,025 ± 0,016 mutaciones por haplotipo por generación., Para el locus trimérico DYS426, donde no se observaron mutaciones en los pedigríes, se utilizó una tasa de regresión basada en un modelo de población logística (0.458 × 10-3 mut/gen, Burgarella y Navascue’s, 2011).
como tasas promedio de mutación por haplotipo HVSI, adoptamos la tasa basada en pedigrí en Howell et al. (2003) basado en 11 estudios de la literatura: 1,06 × 10-2 mut/gen asumiendo un intervalo de generación de 25 años. Es aproximadamente siete veces más alta que la tasa calibrada fósil para el segmento 16,051–16,400 siguiendo a Soares et al. (2009): 1.,42 × 10-3 mut/gen suponiendo un intervalo de generación de 25 años.
análisis de redes
el uso de redes tenía un doble objetivo: muchos de los pasos mutacionales intermedios entre haplotipos, posiblemente oscurecidos por mutación recurrente o por muestreo incompleto, podrían haberse recuperado y considerado en estimaciones de tiempo; la posición de los motivos de haplotipos en el mejor árbol dio una pista de su filogenia.,
Las relaciones mutacionales entre los motivos Y-DNA 12-locus y HVS-I (16,024–16,569 bp) de individuos de ascendencia judía auto-reportada se visualizaron mediante el algoritmo de red de unión mediana implementado en el software Network 4.612 (http://www.fluxus-engineering.com) de acuerdo con Zalloua et al. (2008). Al construir redes, se dio el valor predeterminado (10) a cada sitio HVS-I y se adoptó una puntuación calculada sobre la varianza estimada en cada locus para los datos de Y-STR (puntuación para un locus dado = 10 * varianza total sobre todos los locus/varianza en ese locus dado).,
para y-DNA, seleccionamos solo haplotipos (N = 142) asignados al linaje R1a1a1-M417 y sus subclados aguas abajo que contienen el motivo LMH (16-12-25-10-11-13, Tabla 1), y Haplotipos (n = 73) asignados al linaje J1a2b-P58 y sus subclados aguas abajo que contienen el motivo CMH (14-16-23-10-11-12, cuadro 1). El locus DYS385 no se utilizó porque los alelos no se pueden asignar correctamente a la región duplicada específica, y DYS389II se trató como DYS389b = DYS389II – DYS389I., Los tiempos de coalescencia se estimaron a partir de redes mediante la estadística rho utilizando «pedigríes», así como tasas basadas en «genealogicamente».
para el ADNmt, seleccionamos solo haplotipos que contienen los tres motivos K (16093c 16224C 16311C 16519c 16524g, motif_K1; 16224c 16234t 16311c 16519c, motif_K2; 16224c 16311c 16519c, motif_K3; Tabla 2). Los tiempos de coalescencia se estimaron a partir de redes mediante la estadística rho utilizando tasas basadas en «pedigríes».,
El cálculo de la probabilidad condicional Bayesiana de asignación
El teorema de Bayes se utilizó para calcular la probabilidad condicional de que un individuo pertenezca a la población judía dado que tales individuos llevan un motivo «diagnóstico» de NRY o ADNmt. Tal probabilidad es función de la frecuencia de un haplotipo dado en judíos y no judíos (obtenida de las tablas 3-5) y la proporción de individuos judíos (estimada en el orden de 13 millones) en la población global (7,2 mil millones de personas).

la Tabla 3., Distribution of NRY (typed and predicted upon y-STRs) and mtDNA motifs in the FTDNA public database.
resultados
el análisis de los registros de FtDNA (tablas 3-5) confirmó que ningún motivo genético transmitido a lo largo de la línea materna o paterna es exclusivo de los judíos. Sin embargo, no fue posible extender esta conclusión a subgrupos judíos como levitas y Cohanim porque este estado rara vez se reporta en las entradas de FtDNA., La única excepción es el cromosoma Y «Cohen Zadokites Project» que se une a los descendientes putativos del sobrino de Aarón, Sadoc. Allí, de hecho, pudimos comprobar (tabla S2) que los participantes pertenecían a un total de 6 haplogrupos diferentes con J2, No J1, como modal (63%). No se observó que el haplotipo 12-locus fuera privado para los miembros del proyecto y el J1-eCMH se sumó a solo 4 de los 59 registros con origen conocido (~8%).,
el análisis de distribuciones de haplotipos Y en ramas de árboles parentales confirmó que, aunque hay una clara separación entre la distribución de CMH, eCMH, LMH y eLMH entre poblaciones judías y no judías (resultados de chi cuadrado en la Tabla 4), ningún motivo es diagnóstico de haplogrupos monofiléticos cuando la probabilidad condicional de asignación se estima utilizando la fórmula de Bayes (Figura 1). Incluso los motivos de ADN-Y más resueltos (eLMH y eCMH) se encontraron en dos o más haplogrupos independientes cuyos tiempos superiores de divergencia se estiman hace no menos de 30 K años., Esto podría explicarse por un origen tan profundo y una evolución posterior sin ningún cambio, por los efectos secundarios de la paternidad no reconocida, la conversión génica o, muy probablemente, dada la tasa de cambio de haplotipo (se espera que ocurran alrededor de 24 mutaciones en 30 K años con haplotipos y-STR extendidos) y la alta frecuencia de polifiletismo, por homoplasia.

la Tabla 4. Distribution of motifs in y-DNA haplogrupes (Hg) among 5281 Jews (J) and 57,639 non-Jews (NJ).,

la Figura 1. Estimación del poder diagnóstico de un conjunto de haplotipos modales para asignar una muestra dada a la población judía. Cada línea muestra la probabilidad condicional (fórmula de Bayes) de asignar a un individuo dado que lleva haplotipos modales mtDNA (a) o Ychr (B) a la población judía en función de la proporción de individuos judíos en la muestra. Los puntos rojos muestran la probabilidad posterior obtenida al ingresar la fracción judía estimada de la población mundial.,
vale la pena señalar que las posiciones a lo largo de los árboles de eLMH (central, figura 2a) y eCMH (periférico, figura 2b) sugieren que este último podría no haber estado presente en el grupo inicial de fundadores, sino simplemente ser el resultado de una expansión más reciente.

la Figura 2. Uno de los tres árboles más parsimoniosos construidos por el algoritmo de red de unión mediana., Se utilizaron haplotipos de 12 locus y-STR que contenían el motivo LMH asignado al linaje R1a-M417 (A) y el motivo CMH asignado al linaje J1-P58 (B) junto con sus subclados aguas abajo en individuos que afirmaban ser de ascendencia judía. Los círculos representan haplotipos, con áreas proporcionales al número de individuos que contienen.
el análisis de las distribuciones de haplotipos mitocondriales en judíos indica que, al menos con el nivel actual de resolución de SNP, solo se ha encontrado un motivo (K3) de cada cuatro en diferentes haplogrupos (Tabla 5).,
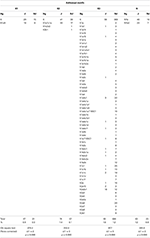
la Tabla 5. Distribution of the four Ashkenazi mtDNA motifs in Haplogroups (Hg) among 2818 Jews (J) and 27,651 non-Jews (NJ).
por otro lado, los hallazgos anteriores enfatizaron una sobre-representación de estos motivos en judíos en comparación con no judíos, así como una acumulación significativa de motivos dentro de ciertos haplogrupos (tablas 4, 5)., Los haplotipos judíos R1a que llevan el motivo levita Ashkenazi LMH (figura 2a) parecen compartir la variante CTS6, cuya TMRCA se estimó entre 1175 ± 341 años atrás (usando la tasa genealógica) y 924 ± 268 años atrás (usando la tasa de línea germinal). Además, los Haplotipos judíos J1 que muestran el motivo Cohanim CMH (figura 2B) parecen compartir la variante Ysc0000234, cuya TMRCA se estimó entre 1255 ± 441 años atrás (usando la tasa genealógica) y 986 ± 346 años atrás (usando la tasa de línea germinal)., Los Haplotipos judíos que llevan los motivos K1, K2 y N son exclusivos, respectivamente, de los haplogrupos K1a9, K1a1b1a y N1b (Tabla 2). Los tiempos de divergencia calculados sobre la variabilidad observada en estos haplogrupos más los haplogrupos K2a2a1 dieron fechas recientes para el ancestro común de todos los motivos de ADNmt, con los de K2 y K3, respectivamente, hace 1370 ± 1241 años y hace 1265 ± 639 años, comparables con los tiempos inferidos para los ancestros comunes de los haplotipos y extendidos.,
discusión
los motivos genéticos hechos de conjuntos de marcadores haploides no recombinantes se han utilizado durante mucho tiempo para rastrear los orígenes judíos putativos de individuos individuales o poblaciones enteras. Tales motivos uni-parentales también se han propuesto para asignar ascendencia en estudios de asociación y casos forenses.
El fracaso de esta práctica es inherente a la naturaleza de la variación genética. Como estimación conservadora podemos esperar una mutación Nueva aproximadamente cada 94 generaciones (28 sustituciones cada 2633 transmisiones, Howell et al.,, 2003) en los linajes mitocondriales y aproximadamente cada tres generaciones (3 × 10-8 mut/site/gen en pedigrees de enraizamiento profundo de más de 10.15 Mb, Xue et al., 2009) en los linajes de NRY.
Por lo tanto, en tales sistemas genéticos cuanto mayor sea el número de marcadores tipificados, menor será la probabilidad de encontrar conjuntos de alelos que coincidan exactamente con la secuencia de los antepasados fundadores., Más específicamente, una mutación ocurriría cada 40-50 transmisiones en haplotipos extendidos y-STR, cada 80-100 transmisiones en haplotipos clásicos 6-locus y-STR y cada 140-150 transmisiones en el segmento 16,024-16,569 de haplotipos HVS-I. Por lo tanto, después de unos pocos miles de años, todos ellos pueden haber mutado más de una vez.
por otro lado, cuanto menor es el número de marcadores tipificados, mayor es la probabilidad de haplotipos idénticos como resultado de homoplasia o de un antiguo linaje común., En otras palabras, la adopción de haplotipos modales como marcadores para ciertos eventos históricos debe tener en cuenta la resolución temporal que ofrece el número de variantes incluidas en los análisis. Los haplotipos definidos por decenas de variantes minimizan el riesgo de identidad por estado (SII), o convergencia, por lo tanto permitiendo un alto poder diagnóstico a expensas de una resolución temporal poco profunda (TMRCA reciente). Los haplotipos definidos por 6 o 12 STRs, como la CMH y la LMH o la eCMH y elmh, permiten una resolución temporal más profunda, pero con un mayor error debido a mutaciones recurrentes., La adopción de secuencias completas de ADNmt podría mejorar la resolución de ciertas filogenias maternas. Sin embargo, de acuerdo con los loci de muchos-Y-STR, esto afectaría necesariamente la compensación entre la potencia para discriminar entre EII y SII y la resolución temporal.
nuestros resultados son una demostración de este argumento: el uso de motivos de haplotipos no recombinantes como marcadores diagnósticos de ascendencia judía demostró ser fuertemente engañoso cuando no está respaldado por el conocimiento en regiones más informativas o secuencias completas., De los motivos que anteriormente se suponía que trazaban ancestros judíos específicos, ninguno resultó idéntico por descendencia (IBD), que se hereda sin modificaciones de un ancestro común. Con pocas excepciones, se observaron motivos, ya fueran del NRY o del ADNmt, en al menos dos linajes independientes, a veces pertenecientes a grupos étnicos con diferentes afiliaciones culturales o geográficas., Para explicar el patrón polifilético de distribución de haplotipos a través de los grupos analizados, prevemos, como la explicación más parsimoniosa, múltiples eventos fundadores y/o reorganización de las piscinas genómicas a través de la larga historia de dispersión y mezcla de comunidades judías desde su fundación.
para hacer un ejemplo, la firma «CMH», en su versión clásica y extendida, se ha observado en muchos haplotipos de habitantes de la Península Arábiga con nombres típicos Árabes, así como en muchos judíos pertenecientes a los haplogrupos J1 y J2., La distribución de la CMHs por etnias y los haplogrupos es tal que sugieren que los genes de las conversiones, las adopciones y ilegítimo paternities podría afectar sólo marginalmente los resultados a menos que ellos eran múltiples y producido cientos de años.
una explicación más fácil es que, entre 7600 y 10.400 años bp (IC95%), el» haplotipo modal de Cohen » fue un haplotipo ancestral para los habitantes históricos de la Península Arábiga., Hace unos 4000 ± 520 años, la población judía establecida llevaba este «haplotipo modal» junto con los futuros árabes, que en ese momento tenían un ancestro común con los futuros judíos (Klyosov, 2010). Alrededor del siglo X D. C., una «CMH reciente» ligeramente modificada se separó de la » CMH más antigua «(en formatos de haplotipos más extendidos), mientras que ambos contenían la firma marcadora 6 de la» CMH», que todavía está presente en muchos haplotipos Árabes. Este «reciente CMH» se convirtió en el haplotipo ancestral de un linaje judío separado aunque reciente dentro del haplogrupo J1., Si se consideran solo haplotipos » CMH » dentro de esta población, se puede identificar un ancestro común que vivió alrededor de 1255-986 años atrás.
centrándose en motivos mitocondriales, la presencia de variantes en sitios de mutación rápida como 16.311 y 16.519 aumenta el potencial de aparición de mutaciones recurrentes en segmentos HVS-I. Esto es particularmente crítico dentro del haplogrupo K., La monofilia y las Tmrca recientes, es decir, las genealogías trazables hasta un único ancestro reciente, podrían invocarse para los motivos K1, K2 y N, mientras que la mezcla y los fundadores múltiples deberían invocarse para el motivo K3 en ausencia de una mejor resolución, en línea con el genoma completo y los datos de re-secuenciación.
Un elemento adicional de incertidumbre es jugado por la elección de la tasa mutacional., Los haplotipos mutan cambiando sus alelos de manera impredecible, y solo se puede predecir un número promedio de mutaciones en un tiempo dado con una cierta probabilidad, basado en constantes de tasa de mutación y en cuán «viejo» es el grupo de haplotipos en términos de un período de tiempo de su ancestro común.
la cuestión de cuál es la constante de tasa de mutación de haplotipo más adecuada para ser aplicada al rastreo de rutas históricas de poblaciones humanas ha sido acaloradamente debatida (ver Soares et al., 2009; Wei et al., 2013) y la reciente disponibilidad de datos de genoma completo y resecuenciación no lo resolvió., Es ampliamente aceptado que los recuentos de mutaciones entre haplotipos divergentes saturan bastante rápidamente debido a mutaciones recurrentes, especialmente en los marcadores STR. También está implícito que las tasas calibradas sobre modelos de ramificación infinita y escalas de tiempo evolutivas inflan TMRCAs de haplotipos que llegaron a divergir en tiempos históricos. Es el caso de la tasa propuesta por Zhivotovsky et al. (2004), que fue utilizado por Hammer y compañeros de trabajo para sostener que la edad de eCMHs es compatible con la fundación del sacerdocio Cohanim (ver crítica en Klyosov, 2009b)., Además, es evidente que las estimaciones basadas en la línea germinal, de padres a hijos o pedigríes de enraizamiento profundo a menudo están respaldadas por estadísticas muy pobres, las mutaciones observadas en algún marcador son muy pocas o ninguna en absoluto.
para complicar la imagen es el concepto de que una tasa constante no existe en el mundo real. Por sí mismo, el número de años en la generación es un valor flotante, depende de los hábitos culturales, las opiniones religiosas, la edad en el nacimiento del niño, la nutrición, la salud y otras condiciones de vida en un territorio dado en un momento dado., Además, la probabilidad de que aparezca una mutación Nueva depende de la estructura de la región genómica donde ocurre y su destino depende en gran medida del tamaño y la demografía de la comunidad a la que pertenece.
como regla general, cuanto más largo sea el tiempo para un ancestro común de un grupo de haplotipos, menos «diagnósticos» será un motivo y estimaciones de tiempo más inciertas., Solo los grupos con antepasados comunes recientes tienen motivos bastante predecibles, como es el caso de los judíos asquenazíes en algunos haplogrupos de ADNmt y subclados Y-R1a expandidos en los últimos mil años, porque no queda mucho tiempo desde el ancestro común, y su haplotipo todavía tiene relativamente pocas mutaciones.
se encontró que todos los motivos propuestos estaban distribuidos de manera desigual entre individuos agrupados de acuerdo con su ascendencia judía y no judía, casi siempre con un enriquecimiento significativo en judíos (tablas 4, 5)., Sin embargo, la composición heterogénea de los haplotipos que los contienen afectó cualquier intento de datar correctamente su origen. La tipificación SNP de mayor resolución y, con suerte, la disponibilidad de secuencias completas, podrían ayudar a refinar la filogenia de dichos marcadores, aclarando finalmente su papel y tiempo desde la fundación de los grupos judíos.,
En conclusión, mientras que la distribución observada de subclados de haplotipos en genomas mitocondriales y cromosomas y no recombinantes podría ser compatible con los eventos fundadores en los últimos tiempos en el origen de grupos judíos como Cohenita, levita, ashkenazita, el polifiletismo sustancial en general, así como su aparición sistemática en grupos no judíos destaca la falta de apoyo para su uso ya sea como marcadores de ascendencia judía o cuentos bíblicos.,
Declaración de conflicto de intereses
los autores declaran que la investigación se realizó en ausencia de relaciones comerciales o financieras que pudieran ser interpretadas como un potencial conflicto de intereses.
reconocimiento
Los autores agradecen a los revisores por sus comentarios críticos y estimulantes.