Introdução
as Sequências do alelo estados ou “motivos”, baseado em polimorfismos no genoma mitocondrial (mtDNA) e o não-recombinantes parte do cromossoma Y (RLE) têm sido propostas para rastreamento Judeu antepassados. Particularmente, Skorecki et al., (1997) first suggested, that the differential distribution of Y-DNA haplotypes based on two markers, the Y Alu polymorphism (YAP) and the Y-STR (Short Tandem Repeat) DYS19, could be a proxy of the patrilineal descent of Cohanim high priests. O haplótipo YAP-/DYS19 B foi reconhecido como o possível haplótipo fundador do sacerdócio judeu. Pouco depois disso, Thomas et al. (1998) refined this hypothesis on the basis of the variability at 6 Y-SNPs (Single Nucleotide Polymorphisms) and 6 Y-STRs., Um haplótipo modal foi encontrado em cromossomos Cohen Y juntamente com um conjunto de haplotipos intimamente relacionados, quer eles pertenciam a comunidades Ashkenazim ou sefarditas, cuja coalescência foi datada a cerca da época do Reino de David (2619-3221 anos atrás). Nebel et al. (2000, 2001) defined the Cohen Modal Haplotype or CMH (Table 1) as a 6-locus Y-STR haplotype belonging to haplogroup Eu10, that is J1-M267 according to the current nomenclature (Y-DNA Haplogroup Tree 2014 Version: 9.70)., Tal motivo resultou em 2-3 mutacional passos de distância de outros Eu10 modal haplótipos observados em língua árabe grupos (MH Galiléia, MH Beduínos, MH Palestinos) e a um passo do parafilético modal haplótipo do Muçulmano Curdos pertencentes a haplogroup Ue9 (J2-M172 na atual nomenclatura). In a subsequent study Thomas and colleagues reported CMH Jewish motifs also from a set of South East African haplotypes (Thomas et al., 2000).
no seu estudo mais recente sobre o assunto, Hammer et al., (2009) have tried to further investigate the Y-DNA evidence of the biblical descent of Cohanim from a single ancestor (the biblical Aaron) by extending the discrimination power of the CMH from 6 – to 12-locus Y-STR haplotypes (eCMH, Table 1). Eles alegaram que a origem da diversidade associada aos cromossomos Cohanim J1-P58 poderia ser datada entre 4280 e 2100 anos atrás.

a Tabela 1. NRY haplotype motifs.,
os Haplotipos modais de levita (LMH e eLMH, Quadro 1) foram propostos por Behar et al. (2003) as alternative Y-STR modal haplotypes within the R1a-M17 haplogroup variation of Ashkenazim Levites, partly shared with the Eastern Europeans. Esta descoberta desencadeou a hipótese da origem do Iídiche a partir de uma língua eslava (Sorbiana) e a introgressão dos cromossomos Y Cazarianos na formação inicial dos judeus Ashkenazi há cerca de 1000 anos., No entanto, análises de resequenciamento encontraram vários eventos fundadores entre os levitas Ashkenazi dentro da R1a demonstrando que um determinado sub-clado, o M582, seria uma assinatura de uma origem do Oriente Próximo nos tempos pré-diáspora (Rootsi et al., 2013).Thomas et al. (2002) found signals of much stronger founding events in female-specific (mtDNA) lineages of different Jewish communities than in corresponding male-specific lineages., Eles encontraram pelo menos 8 haplotipos modais (frequência >10%) na região HVS-I do DNA mitocondrial em 8 comunidades geograficamente separadas de judeus, enquanto que não foi observada diferenciação em haplotipos Y de populações judaicas e hospedeiras.usando uma análise de alta resolução dos haplogroups K E N, Behar e seus colaboradores identificaram quatro linhagens fundadoras do mtDNA como a fonte matrilínea de cerca de 40% da população atual de judeus Ashkenazi (Behar et al., 2006)., Tais linhagens foram descritas como originárias do oriente médio por volta de 2100 anos atrás “provavelmente de um pool Hebraico/Levantino mtDNA.”No entanto, a origem matrilínea complexa dos Ashkenazi parece ter sido melhor representada por Costa et al. (2013), que revelou que a grande maioria das linhagens maternas Ashkenazi foram o resultado de eventos de mistura reiterada na Europa. No que se refere aos potenciais indicadores demográficos do cenário supra, Behar et al. (2004) and Guha et al., (2012) têm reivindicado uma forte deriva genética para contribuir para a anormalmente alta frequência de alelos da doença recessiva e baixa diversidade mtDNA e Y-DNA nas populações Ashkenazi. Por outro lado, a literatura recente baseada em análises genómicas (Atzmon et al., 2010; Behar et al., 2010; Bray et al., 2010) destacou o papel decisivo da mistura na formação da atual piscina de DNA Judaico.nos últimos anos, empresas genéticas Genealógicas têm recrutado dezenas de milhares de voluntários que aceitaram partilhar os resultados dos seus testes genéticos em repositórios públicos., O maior banco de dados de ancestralidade disponível até à data é o Family Tree DNA archive (FtDNA), incluindo mais de 506 K registros para Y-DNA e cerca de 180 K registros para mtDNA dividido em quase 8 K projetos, onde os participantes podem compartilhar seu próprio perfil de DNA para traçar um patrimônio comum pelo sobrenome, linhagem ou geografia. Fazendo uso de tal recurso valioso, construímos uma grande base de dados de perfis de mtDNA e Y-DNA disponível a partir de >600 FtDNA projetos., Exploramos o banco de dados para examinar a variabilidade associada aos motivos genéticos propostos pela literatura em voluntários que reivindicam uma ascendência judaica e em grupos não-judeus de origem europeia, africana ou do Oriente Médio.
we aimed to providing an updated experimental background by which to argument avances and pitfalls one may encounter when using haplotype motifs as diagnostic markers of Jewish ancestry.,
materiais e métodos
bases de Dados construir
os haplotipos Y-STR e Y-SNPs foram descarregados do navegador de cromossomas y Da Árvore Genealógica (https://www.familytreedna.com/projects.aspx). To match reference motifs (Behar et al., 2003) com directrizes de nomenclatura normalizadas (Gusmão et al., 2006) allele states at DYS439 were corrected subtracting 4 reprises. Os haplotipos HVS-I e mtDNA SNPs foram descarregados do navegador de cromossomas DNA mt Da Árvore Genealógica (https://www.familytreedna.com/projects.aspx).,
registros (https://www.familytreedna.com/projects.aspx) foram divididos pela primeira vez em projetos destinados a reconstruir explicitamente ancestralidade judaica e outros projetos. Em segundo lugar, filtramos dentro e entre projetos esses registros com números de kit duplicados e origens incertas. Quando não estão disponíveis, as associações entre haplotipos e SNPs foram feitas por número de kit., Por último, nós removemos os haplótipos que não se encaixam um duplo critério: RLE-haplótipos – ser digitados em um mínimo de 12 Y-loci (o conjunto de “extended” motivos) e atribuído a haplogrupos por directos SNP digitação ou sobre previsões com base em ≥25-locus STR perfis; dnamt-haplótipos—para ser sequenciado pelo menos para HVS-I sites 16,024–16,569 e atribuído a haplogrupos por digitação direta de diagnóstico de SNPs. Obtivemos uma grelha final de 62.920 Y-DNA e 30.469 mtDNA records. As bases de dados foram pesquisadas por motivos de 6 e 12 Y-STR (judeus e árabes) e por 4 motivos HVS-i Ashkenazi (Tabelas 1, 2).,

Tabela 2. mtDNA haplotype motifs.
uma limitação dos arquivos genealógicos genéticos publicamente disponíveis é a auto-atribuição dos participantes a categorias sociais/étnicas, como é o caso da descendência judaica. Outra é a relação putativa entre os participantes dentro do mesmo grupo étnico., Embora o primeiro limite seja difícil de determinar, e não possam ser excluídas fontes de erro como adopções e paternidade ilegítimas, uma análise preliminar parece afectar apenas marginalmente as estatísticas resumidas apresentadas no presente artigo relativamente às de relatórios científicos anteriores. Registros compartilhando um ancestral comum foram 12 de 3903 conhecidos (0,3%) em projetos de ascendência judaica e 606 de 48.006 conhecidos (1,5%) nos outros projetos.,
Cálculo do Haplótipo Taxas de Mutação
empregamos o seguinte pedigree taxas baseadas em:
Média das taxas de mutação por Y-haplótipos foram obtidos de acordo com o “genealógica” (método de Klyosov, 2009a; Rozhanskii e Klyosov, 2011) após a calibração de volta mutações descritas, e “germinal”, estima a combinação de pai-para-filho pares de dados da literatura (29 estudos revisados por Burgarella e Navascue s, De 2011, além de Ballantyne et al., 2010).,
No primeiro caso, a inclinação da curva de calibração para o 12-locus de STRs de eCMH e eLMH motivos se encaixa bem para um valor de a taxa de mutação constante de 0.00166 mutação por marcador por condicional, geração de 25 anos, que é 0,020 mutações por haplótipo a cada 25 anos.
neste último caso, o número de mutações observadas em um total de 126,873 meioses (Tabela S1) apresentou uma taxa média 2.113 × 10-3 (± 1.369 × 10-3) mutações por marcador por geração, que é de 0,025 ± 0.016 mutações por haplótipo por geração., Para o trimérico locus DYS426, onde não foram observadas mutações entre os pedigrees, foi utilizada uma taxa de regressão baseada em um modelo de população logística (0.458 × 10-3 mut/gen, Burgarella e Navascue, 2011).como taxas médias de mutação por hvsi-haplótipo, adoptámos a taxa combinada baseada em pedigree em Howell et al. (2003) based on 11 studies from the literature: 1,06 × 10-2 mut/gen assumpting a generation interval of 25 years. É cerca de sete vezes maior do que a taxa calibrada fóssil para o segmento 16.051–16.400 seguindo Soares et al. (2009): 1.,42 × 10-3 mut / gen assumindo um intervalo de geração de 25 anos.
Rede Análises
O uso de redes tinham um duplo objetivo: muitos de entre-os haplótipos intermediário mutacional etapas, possivelmente obscurecida pela recorrente mutação ou incompleta de amostragem, poderia ter sido recuperados e levados em tempo estimativas; a posição do haplótipo motivos sobre a melhor árvore deu uma pista de sua filogenia.,
Mutacional relações entre o Y-DNA de 12 locus motivos e HVS-I (16,024–16,569 bp) motivos de indivíduos de auto-relato de ascendência Judaica foram visualizados por meio da mediana de ingressar na rede algoritmo implementado na Rede 4.612 de software (http://www.fluxus-engineering.com) de acordo com Zalloua et al. (2008). Ao construir redes, o valor padrão (10) foi dado a cada site HVS-I e uma pontuação calculada sobre a variância estimada em cada locus foi adotado para os dados Y-STR (pontuação para um locus dado = 10 * variância total sobre todo o loci/variância naquele locus dado).,
Para Y-DNA, foram selecionados apenas os haplótipos (N = 142) atribuído ao R1a1a1-M417 linhagem e a sua jusante subclades contendo o LMH motivo (16-12-25-10-11-13, Tabela 1), e os haplótipos (N = 73) atribuído ao J1a2b-P58 linhagem e a sua jusante subclades contendo o CMH motivo (14-16-23-10-11-12, Tabela 1). O locus DYS385 não foi usado porque alelos não podem ser corretamente atribuídos à região duplicada específica, e DYS389II foi tratado como DYS389b = DYS389II – DYS389I., Os tempos de coalescência foram estimados a partir de redes pela rho statistic usando “pedigrees” – bem como taxas baseadas na genealogia.
Para o dnamt, foram selecionados apenas os haplótipos, contendo os três K motivos (16093C 16224C 16311C 16519C 16524G, motif_K1; 16224C 16234T 16311C 16519C, motif_K2; 16224C 16311C 16519C, motif_K3; Tabela 2). Os tempos de coalescência foram estimados a partir de redes pela rho statistic usando taxas baseadas em “pedigrees”.,
Cálculo Bayesiano de Probabilidade Condicional de Atribuição
teorema de Bayes foi usado para calcular a probabilidade condicional de um indivíduo pertencer a população Judaica, visto que tais indivíduos carrega um “diagnóstico” RLE ou dnamt motivo. Tal probabilidade é função da frequência de um dado haplótipo em Judeus e não-judeus (obtido a partir das tabelas 3-5) e da proporção de indivíduos judeus (estimado em cerca de 13 milhões) na população global (7,2 bilhões de pessoas).

a Tabela 3., Distribuição de NRY (tipado e previsto em Y-STRs) e mtDNA motifs na base de dados pública FTDNA.
resultados
a análise dos registos FtDNA (quadros 3-5) confirmou que nenhum motivo genético transmitido ao longo da linha materna ou paterna é exclusivo dos judeus. No entanto, não foi possível estender esta conclusão a subgrupos judaicos como levitas e Cohanim porque este status raramente é auto-relatado em entradas FtDNA., A única exceção é o Y cromossomo “Cohen Zadokites Project”, que se junta aos descendentes putativos do sobrinho de Aaron, Zadok. Lá, de fato, poderíamos verificar (tabela S2) que os participantes pertenciam a um total de 6 haplogroups diferentes com J2, não J1, como modal (63%). No 12-locus haplotype was observed to be private to the members of the Project and the J1-eCMH somed to only 4 out of the 59 records with known origin (~8%).,
A análise de Y haplótipo distribuições em parental ramos de árvore, que confirmou que, embora exista uma separação clara entre a distribuição de CMH, eCMH, LMH, e eLMH entre Judeus e não-Judeus populações (qui-quadrado resultados na Tabela 4), não há motivo é o diagnóstico de monofilético haplogrupos quando a probabilidade condicional de atribuição é estimado utilizando-se a fórmula de Bayes (Figura 1). Mesmo os motivos Y-DNA mais resolvidos (eLMH e eCMH) foram encontrados em dois ou mais haplogrupos independentes cujos tempos superiores de divergência são estimados não menos de 30 K anos atrás., Isto pode ser explicado por uma origem tão profunda e subsequente evolução sem qualquer alteração, pelos efeitos colaterais da paternidade não reconhecida, conversão genética ou, muito provavelmente, dada a taxa de mudança de haplótipo (cerca de 24 mutações são esperadas para ocorrer em 30 K anos em haplotipos Y-STR estendidos) e a alta frequência do polifiletismo, por homoplasia.
Tabela 4. Distribuição de motivos em haplogroups de DNA Y (Hg) entre 5281 judeus (J) e 57.639 não-judeus (NJ).,

a Figura 1. Estimation of diagnostic power of a set of modal haplotypes to assign a given sample to the Jewish population. Cada linha mostra a probabilidade condicional (fórmula Bayes) de atribuir um determinado indivíduo Carregando haplotipos modais mtDNA (a) ou Ychr (B) para a população judaica em função da proporção de indivíduos judeus na amostra. Pontos vermelhos mostram a probabilidade posterior obtida ao inserir a fração judaica estimada da população mundial.,
vale lembrar que as posições ao longo das árvores de eLMH (central, Figura 2A) e eCMH (periférico, Figura 2B) sugerem que este último pode não ter sido presente no início da piscina de fundadores, mas simplesmente o resultado da mais recente expansão.

Figura 2. Uma das três árvores mais parsimónicas construídas pelo algoritmo de ligação mediana., Usámos haplotipos y-STR de 12 locus contendo o motivo LMH atribuído à linhagem R1a-M417 (a) e o motivo CMH atribuído à linhagem J1-P58 (B), juntamente com as suas subclades a jusante em indivíduos que reivindicam uma descendência judaica. Os círculos representam haplotipos, com áreas proporcionais ao número de indivíduos que contêm.
a análise das distribuições de haplotipos mitocondriais em Judeus indica que, pelo menos com o nível atual de resolução SNP, apenas um motivo (K3) de quatro foi encontrado em diferentes haplogrupos (Tabela 5).,
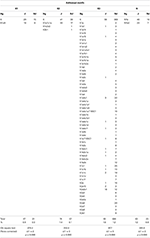
a Tabela 5. Distribuição dos quatro motivos Ashkenazi mtDNA em haplogroups (Hg) entre 2818 judeus (J) e 27.651 não-judeus (NJ).
por outro lado, os resultados acima enfatizaram uma sobre-representação destes motivos Em judeus quando comparados com não-judeus, bem como uma acumulação significativa de motivos dentro de certos haplogroups (tabelas 4, 5)., O R1a Judeu haplótipos carregando o Ashkenazi Levita motivo LMH (Figura 2A) parecem compartilhar a CTS6 variante, cujo TMRCA foi estimada entre 1175 ± 341 anos atrás (usando o genealógica taxa) e 924 ± 268 anos atrás (usando o germe de taxa de linha). Assim, o J1 Judeu haplótipos exibir os Cohanim motivo de CMH (Figura 2B) parecem compartilhar a YSC0000234 variante, cujo TMRCA foi estimada entre 1255 ± 441 anos atrás (usando o genealógica taxa) e 986 ± 346 anos atrás (usando o germe de taxa de linha)., Os haplotipos judeus que transportam os motivos K1, K2 E N são exclusivos, respectivamente, dos haplogrupos K1a9, K1a1b1a e N1b (Quadro 2). Divergência vezes calculada sobre a variabilidade observada nestes haplogrupos mais o K2a2a1 haplogrupos deu datas recentes para o ancestral comum de todos os haplogrupos de motivos, com aqueles para K2 e K3, respectivamente, 1370 ± 1241 anos atrás e 1265 ± 639 anos atrás, comparáveis com os tempos inferida para os ancestrais comuns a extensa Y haplótipos.,
discussão
motivos genéticos feitos de conjuntos de marcadores haplóides não recombinantes têm sido usados há muito tempo para rastrear as origens judaicas putativas de indivíduos individuais ou populações inteiras. Tais motivos uni-parentais também foram propostos para atribuir ascendência em estudos de associação e casos forenses.a falha desta prática é inerente à natureza da variação genética. Como estimativa conservadora, podemos esperar uma nova mutação a cada 94 gerações (28 substituições a cada 2633 transmissões, Howell et al.,, 2003) in the mitochondrial lineages and about every three generations (3 × 10-8 mut/site/gen in deep-rooting pedigrees over 10.15 Mb, Xue et al., 2009) in the NRY lineages.
portanto, em tais sistemas genéticos, quanto maior o número de marcadores tipados, menor a probabilidade de encontrar conjuntos alélicos que correspondem exatamente à sequência dos ancestrais fundadores., Mais especificamente, uma mutação ocorreria a cada 40-50 transmissões em haplotipos y-STR estendidos, cada 80-100 transmissões em haplotipos y-STR clássicos 6-locus e cada 140-150 transmissões no segmento 16,024–16,569 haplotipos HVS-I. Assim, após alguns milhares de anos, todos eles podem ter sofrido mutações mais de uma vez.por outro lado, quanto menor o número de marcadores tipados, maior a probabilidade de haplotipos idênticos como resultado de homoplasia ou de uma antiga descendência comum., Por outras palavras, a adopção de haplotipos modais como marcadores de certos acontecimentos históricos deve ter em conta a resolução temporal proporcionada pelo número de variantes incluídas nas análises. Y haplotipos definidos por dezenas de variantes minimizam o risco de identidade por estado (IBS), ou convergência, permitindo, portanto, alta potência de diagnóstico às custas de uma resolução temporal superficial (TMRCA recente). Os haplotipos definidos por 6 ou 12 STRs, como o CMH e LMH ou o eCMH e eLMH, permitem uma resolução temporal mais profunda, mas com um erro aumentado devido a mutações recorrentes., A adoção de sequências inteiras de mtDNA poderia melhorar a resolução de certas filogenias maternas. De acordo com o many-Y-STR loci, no entanto, isso afetaria necessariamente a troca entre o poder de discriminar entre IBD e IBS e resolução temporal.
nossos resultados são uma demonstração deste argumento: o uso de motivos haplotipos não-recombinantes como marcadores diagnósticos de ancestralidade judaica foi mostrado ser fortemente enganador quando não suportado pelo conhecimento em regiões mais informativas ou sequências inteiras., Dos motivos anteriormente assumidos para traçar ancestrais judeus específicos, nenhum resultou idêntico pela descendência (IBD), que é herdada sem modificações de um ancestral comum. Com poucas exceções, motivos, sejam do NRY ou do mtDNA, foram observados em pelo menos duas linhagens independentes, algumas vezes pertencentes a grupos étnicos com diferentes afiliações culturais ou geográficas., Para explicar o polyphyletic padrão de distribuição de haplótipos entre os grupos analisados, prevê-se, como o mais parcimonioso explicação, vários fundador eventos e/ou reformulações de genômica piscinas através da longa história de dispersão e mistura de comunidades Judaicas desde a sua fundação.
para dar um exemplo, a assinatura” CMH”, em sua versão clássica e estendida, tem sido observada em muitos haplotipos de habitantes da Península Arábica com nomes árabes típicos, bem como em muitos judeus pertencentes a haplogroups J1 e J2., A distribuição de CMS por etnias e haplogrupos é tal que sugere que a conversão de genes, adopções e paternidade ilegítimas poderiam afetar apenas marginalmente os resultados, a menos que fossem múltiplos e ocorressem principalmente há centenas de anos.
uma explicação mais fácil é que, entre 7600 e 10.400 anos bp (95% IC), o “haplótipo Modal Cohen” foi um haplótipo ancestral para os habitantes históricos da Península Arábica., Cerca de 4000 ± 520 anos atrás, a população judaica estabelecida carregava este “haplótipo modal” juntamente com os futuros árabes, que naquela época tinha um ancestral comum com os futuros judeus (Klyosov, 2010). Por volta do século X, um “CMH recente” ligeiramente modificado do “antigo CMH” (em formatos de haplotipos mais extensos), enquanto ambos continham a assinatura de 6 marcadores do “CMH”, que ainda está presente em muitos haplotipos Árabes. Este “recente CMH” tornou-se o haplótipo ancestral para uma linhagem judaica separada, embora recente, dentro do haplogroup J1., Se considerarmos apenas haplotipos ” CMH ” dentro desta população, um ancestral comum que viveu por volta de 1255-986 anos atrás pode ser identificado.com foco em motivos mitocondriais, a presença de variantes em locais de mutação rápida como 16.311 e 16.519 aumenta o potencial para a ocorrência de mutações recorrentes em segmentos HVS-I. Isto é particularmente crítico no haplogrupo K., Monofilias e recentes TMRCAs, ou seja, genealogias rastreáveis até um único ancestral recente, poderiam ser invocadas para os motivos K1, K2 E N, enquanto a mistura e múltiplos fundadores devem ser invocados para o motivo K3 na ausência de uma melhor resolução, de acordo com o genoma completo e os dados de re-sequenciação.
um elemento adicional de incerteza é jogado pela escolha da taxa mutacional., Haplotype muda mudando seus alelos imprevisivelmente, e apenas um número médio de mutações ao longo de um determinado tempo pode ser previsto com uma certa probabilidade, com base em constantes de taxa de mutação e em como “velho” é o grupo de haplotypes em termos de um período de tempo de seu ancestral comum.
a questão de qual é a constante de taxa de mutação de haplotipos mais adequada a ser aplicada ao traçado das vias históricas das populações humanas foi muito debatida (ver Soares et al., 2009; Wei et al., 2013) e a recente disponibilidade de dados completos sobre genoma e resequenciamento não resolveu o problema., É amplamente aceito que as contagens de mutação entre haplotipos divergentes saturam muito rapidamente por causa de mutações recorrentes, especialmente em Marcadores STR. Também está implícito que as taxas calibradas em modelos ramificados infinitos e escalas evolutivas inflacionam TMRCAs de haplotipos que vieram a divergir em tempos históricos. É o caso da taxa proposta por Zhivotovsky et al. (2004), que foi usado por Hammer e colegas para sustentar que a idade dos eCMHs é compatível com a fundação do sacerdócio Cohanim (ver crítica em Klyosov, 2009b)., Além disso, é evidente que a linha germinal, de pais para filhos ou as estimativas baseadas em pedigrees enraizadas são muitas vezes apoiadas por Estatísticas muito pobres, sendo as mutações observadas em algum marcador muito poucas ou nenhumas.
para complicar a imagem é o conceito de que uma taxa constante não existe no mundo real. Por si só, o número de anos na geração é um valor flutuante, depende de hábitos culturais, visões religiosas, idade no nascimento dos filhos, nutrição, saúde e outras condições de vida em um determinado território em um determinado momento., Além disso, a probabilidade de uma nova mutação aparecer depende da estrutura da região genómica onde ela acontece e o seu destino depende em grande medida do tamanho e da demografia da comunidade a que pertence.
Como regra, o tempo de vida mais longo para um ancestral comum de um grupo de haplotipos, menos “diagnósticos” um motivo e estimativas de tempo mais incertas., Apenas grupos com ancestrais comuns recentes têm motivos bastante previsíveis, como é o caso dos judeus Ashkenazi em alguns haplogrupos mtDNA e sub-clados Y-R1a expandiram nos últimos mil anos, porque não há muito tempo desde o ancestral comum, e seu haplótipo ainda está em torno de ter relativamente poucas mutações.todos os motivos propostos foram encontrados de forma desigual entre os indivíduos agrupados de acordo com sua ascendência judaica e não judaica auto-relatada, quase sempre com um enriquecimento significativo nos judeus (tabelas 4, 5)., No entanto, a composição heterogênea dos haplotipos que os contêm afetou qualquer tentativa de datação correta de sua origem. Higher resolution SNP typing and, hopefully, the availability of full sequences, might help refining the phylogeny of such markers, ultimately clarifying their role and time from the foundation of the Jewish groups.,
Em conclusão, embora a observada distribuição de sub-subtipos de haplótipos em mitocondrial e o cromossomo Y não-recombinante genomas podem ser compatível com o fundador eventos nos últimos tempos na origem de grupos Judaicos como Cohenite, Levita, Ashkenazite, em geral, a substancial polyphyletism bem como a sua ocorrência sistemática de grupos não Judeus destaca a falta de suporte para utilizá-los como marcadores de ascendência Judaico ou contos Bíblicos.,
Declaração de conflito de interesses
os autores declaram que a investigação foi realizada na ausência de quaisquer relações comerciais ou financeiras que possam ser interpretadas como um potencial conflito de interesses.
reconhecimento
os autores gostariam de reconhecer os revisores pelos seus comentários críticos e estimulantes.material suplementar