la mutación genética es un riesgo importante para las células vivas. El daño directo al ADN o los errores en los procesos que generan ARN mensajero (ARNm) a partir de la plantilla de ADN pueden introducir mutaciones, con consecuencias potencialmente dañinas. Las mutaciones «sin sentido» son particularmente problemáticas: están asociadas con muchas enfermedades hereditarias genéticamente, como el trastorno sanguíneo β-talasemia, y son comunes en el cáncer (Bhuvanagiri et al., 2010)., Para reducir el impacto de estas mutaciones, las células eucariotas han desarrollado métodos conocidos como decaimiento mediado por tonterías (o NMD para abreviar) para destruir moléculas de ARNm mutantes. Ahora, en Elife, Ligang Wu y compañeros de trabajo en los Institutos de Shanghai para las ciencias biológicas-incluyendo a ya Zhao como el primer autor—reportan un nuevo mecanismo de descomposición mediada por tonterías que es impulsado por microRNAs, una importante familia de moléculas reguladoras(Zhao et al., 2014).,
la expresión génica implica la transcripción, en la que el ARNm se copia de una plantilla de ADN, seguida de la traducción, en la que una máquina molecular llamada ribosoma interpreta la secuencia de un ARNm para hacer la proteína que codifica. Las señales dentro del ARNm llamadas codones stop le dicen al ribosoma cuándo dejar de traducir el ARNm (figura 1a). Las mutaciones sin sentido introducen un codón de parada’ aguas arriba ‘ de la señal correcta para que la traducción se detenga pronto y se produzca una proteína truncada., Los truncamientos pueden interferir con la función normal de la proteína de varias maneras: si se pierde una región reguladora, la proteína puede ser sobre-activa; las proteínas no funcionales acortadas también pueden desplazar versiones funcionales de la misma proteína de complejos multiproteicos.
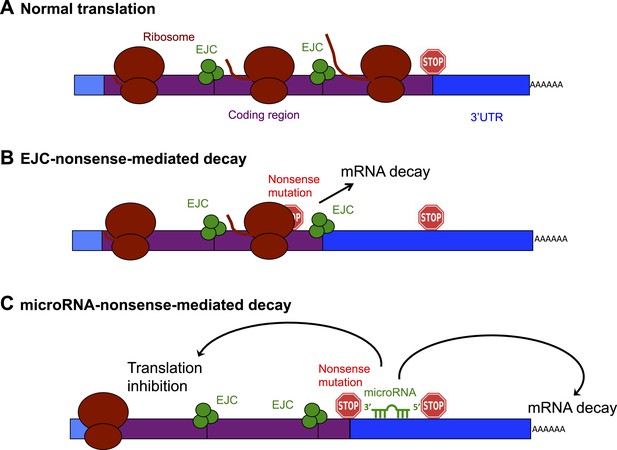
diagrama esquemático que muestra el complejo de unión de exones (EJC) y los mecanismos de desintegración mediada por el microRNA (NMD).,
(A) en una molécula normal de ARNm (rectángulo azul y púrpura), el ribosoma (óvalos marrones) escanea a lo largo de la región codificante (púrpura), y traduce una proteína (línea marrón); este proceso se detiene cuando el ribosoma encuentra un codón de parada (signo de parada). El ARNm después del codón de parada se llama la Región 3 ‘ sin traducir (3’UTR, azul), y no codifica una proteína. (B) en la descomposición mediada por EJC-nonsense, si una mutación sin sentido se encuentra más de 50 nucleótidos aguas arriba de un complejo de unión de exones (EJC; círculos verdes), esto se reconoce como aberrante y el ARNm se degrada. C) Zhao y otros, han descubierto un nuevo método de descomposición mediada por tonterías que depende de las acciones reguladoras de las moléculas de microRNA. La región de codificación aguas abajo de una mutación sin sentido se comporta como 3’UTR. Si un sitio de unión de microRNA está presente en esta región, el microRNA se une a él, lo que inhibe la traducción y conduce a que la molécula de ARNm se degrade.,
el método mejor entendido de decaimiento mediado por tonterías se basa en un proceso conocido como empalme, que elimina segmentos de un ARNm a medida que se está construyendo y pega las partes restantes, llamadas exones, para hacer el ARNm maduro. Donde se unen dos exones, la célula deposita proteínas que forman un complejo de unión de exones (EJC) en la molécula de ARNm. Cuando este ARNm maduro está siendo traducido, el ribosoma mide la distancia entre el EJC y el codón stop., Si un EJC se encuentra más de 50 nucleótidos después (o ‘aguas abajo’ de) un codón de parada, la célula reconoce esto como aberrante y destruye el ARNm (Popp y Maquat, 2013; figura 1B).
la región del ARNm aguas abajo del codón de parada se conoce como la Región 3 ‘ NO traducida (3’UTR). Aunque esta región no codifica ninguna proteína, es importante para regular la actividad de la molécula de ARNm. Los MicroRNAs son moléculas cortas de ARN no codificantes (es decir, no codifican para proteínas) que se unen a los sitios 3’UTR de una manera secuencial específica., La Unión de microRNAs puede inhibir la traducción y fomentar la degradación de una molécula de ARNm, y es muy importante para controlar cómo y cuándo se expresan los genes en los eucariotas (Fabian y Sonenberg, 2012).
Zhao et al. razonó que si un ARNm contiene una mutación sin sentido, la región de codificación aguas abajo de la mutación se convertirá esencialmente en un 3’UTR (Zhao et al., 2014). Este 3’UTR extendido podría contener sitios de unión para microRNAs, que se unirían y regularían la molécula de ARN mensajero. Zhao et al., utilizó una serie de experimentos elegantes para confirmar esta hipótesis, y, al hacerlo, identificó un nuevo método de decaimiento mediado por tonterías que es dirigido por microRNAs (figura 1C). Primero, Zhao et al. inserted a microRNA binding site into the coding region of an mRNA. Esto no tuvo ningún efecto en el ARNm, confirmando la opinión prevaleciente de que la regulación del microRNA no ocurre de manera eficiente en las regiones codificantes (Gu et al., 2009). Sin embargo, cuando Zhao et al. introdujo un codón sin sentido para que el sitio del microRNA ahora se ubicara en el 3’UTR, los microRNAs podrían unirse a él, lo que indujo la descomposición del ARNm(Zhao et al., 2014).,
Zhao et al. pasó a mostrar que los microRNAs también influyen en la decadencia mediada por tonterías en los ARNm que ocurren naturalmente. Se centraron en APC, un gen supresor de tumores que a menudo se encuentra que contiene mutaciones sin sentido en tumores de cáncer colorrectal. El gen APC fue una opción particularmente buena para este estudio, ya que la mayoría de sus mutaciones sin sentido ocurren en un punto caliente dentro del último exón, por lo que no son reconocidas por el método complejo de unión de exones de descomposición mediada por tonterías (Miyoshi et al., 1992). Sin embargo, Zhao et al., se encontró que mutaciones sin sentido en esta región exponen sitios de unión de microRNA dentro del recién formado 3’UTR, lo que conduce a la descomposición del ARNm copiado del gen APC. Finalmente, Zhao et al. se utilizaron genes reporter basados en el gen supresor de tumores asociado al cáncer de mama BRCA1, que es sensible a la forma EJC de descomposición mediada por tonterías, para mostrar que el complejo de unión de exones y los mecanismos de microRNA de descomposición mediada por tonterías pueden funcionar en el mismo ARNm (Zhao et al., 2014).
los hallazgos de Zhao et al., revela una nueva función intrigante para los microRNAs y un nuevo mecanismo de decadencia mediada por tonterías. También plantean varias cuestiones importantes. ¿Cuál es la contribución relativa de EJC y microRNA-driven nonsense-mediated decaimiento en las células? En contraste con la descomposición mediada por el sinsentido de la EJC, la regulación del microRNA tiende a ser bastante débil (Leung y Sharp, 2010). Sin embargo, múltiples sitios de microRNA funcionan cooperativamente, por lo que el nivel de descomposición mediada por microRNA sin sentido es probable que esté fuertemente influenciado por el número y la identidad de los sitios de microRNA revelados por una mutación sin sentido., Además, la expresión de microRNA está altamente regulada durante el desarrollo y en ciertos tejidos, así como en los cánceres, lo que sugiere que la descomposición mediada por microRNA mediada por tonterías también es probable que esté estrechamente regulada.
también se han descubierto otros métodos de descomposición mediada por tonterías independientes de la EJC (Metze et al., 2013), entonces, ¿cómo interactúa el método microRNA con estos mecanismos alternativos? Al regular las proteínas involucradas en la desintegración mediada por el complejo de unión de exones, los microRNAs pueden influir en cómo procede la desintegración (Bruno et al., 2011)., Además, un nuevo estudio ha demostrado que la Unión de microRNA puede crear un objetivo adecuado para la descomposición mediada por tonterías EJC cambiando la forma en que el ribosoma lee una molécula de ARNm específica (CCR5, que codifica una proteína utilizada por muchas formas de VIH para invadir las células; Belew et al., 2014). En general, parece que la complejidad de cómo la célula trata con mutaciones sin sentido y cómo los microRNAs están involucrados solo está comenzando a revelarse.