une mutation Génétique est un risque majeur pour les cellules vivantes. Des dommages directs à L’ADN ou des erreurs dans les processus qui génèrent L’ARN messager (ARNm) à partir du modèle D’ADN peuvent introduire des mutations, avec des conséquences potentiellement néfastes. Les mutations « absurdes » sont particulièrement problématiques: elles sont associées à de nombreuses maladies génétiquement héréditaires, telles que le trouble sanguin β-thalassémie, et sont fréquentes dans le cancer (Bhuvanagiri et al., 2010)., Pour réduire l’impact de ces mutations, les cellules eucaryotes ont développé des méthodes connues sous le nom de désintégration Non-sensée (ou NMD pour faire court) pour détruire les molécules d’ARNm mutantes. Maintenant, dans eLife, Ligang Wu et ses collègues des instituts de Shanghai pour les Sciences biologiques-y compris Ya Zhao en tant que premier auteur—rapportent un nouveau mécanisme de désintégration non-sens qui est entraîné par les microARN, une importante famille de molécules régulatrices (Zhao et al., 2014).,
l’expression génique implique la transcription, dans laquelle l’ARNm est copié à partir d’un modèle D’ADN, suivie d’une traduction, dans laquelle une machine moléculaire appelée le ribosome interprète la séquence d’un ARNm pour fabriquer la protéine qu’il Code. Les signaux à l’intérieur de l’ARNm appelés codons stop indiquent au ribosome quand arrêter de traduire l’ARNm (Figure 1A). Les mutations non-sens introduisent un codon d’arrêt « en amont » du signal correct de sorte que la traduction est arrêtée tôt et une protéine tronquée est faite., Les troncations peuvent interférer avec la fonction normale des protéines de diverses manières: si une région régulatrice est perdue, la protéine peut être sur-active; les protéines non fonctionnelles raccourcies peuvent également déplacer les versions fonctionnelles de la même protéine des complexes multi-protéines.
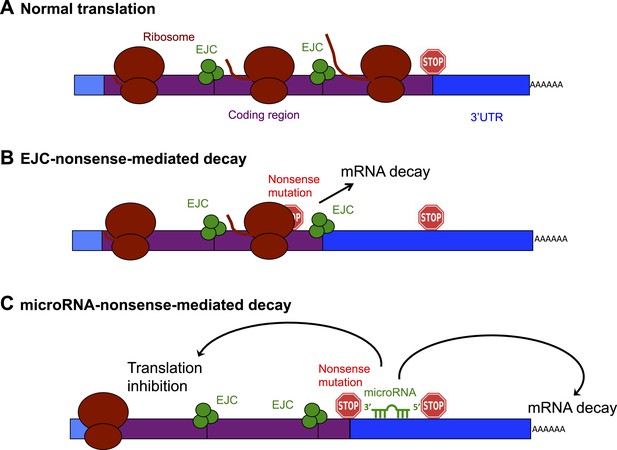
diagramme schématique montrant le complexe de jonction d’exons (EJC) et les mécanismes pilotés par les microARN de désintégration non-sens (NMD).,
(a) dans une molécule d’ARNm normale (rectangle bleu et violet), le ribosome (ovales bruns) scanne le long de la région codante (violet) et traduit une protéine (ligne brune); ce processus s’arrête lorsque le ribosome rencontre un codon stop (signe stop). L’ARNm après le codon stop est appelé la région non traduite 3′ (3’UTR, bleu), et ne Code pas une protéine. (B) dans la désintégration médiée par EJC-nonsense, si une mutation non-sens est située à plus de 50 nucléotides en amont d’un complexe de jonction d’exon (EJC; cercles verts), cela est reconnu comme aberrant et l’ARNm est dégradé. C) Zhao et consorts, ont découvert une nouvelle méthode de désintégration non-sens qui dépend des actions de régulation des molécules de microARN. La région codante en aval d’une mutation non-sens se comporte comme 3’UTR. Si un site de liaison de microARN est présent dans cette région, le microARN s’y lie, ce qui inhibe la traduction et conduit à la dégradation de la molécule d’ARNm.,
la méthode la mieux comprise de désintégration Non-sensée repose sur un processus connu sous le nom d’épissage, qui supprime les segments d’un ARNm pendant sa construction et colle les parties restantes—appelées exons—ensemble pour faire l’ARNm mature. Lorsque deux exons sont joints, la cellule dépose des protéines qui forment un complexe de jonction d’exons (EJC) sur la molécule d’ARNm. Lorsque cet ARNm mature est traduit, le ribosome mesure la distance entre L’EJC et le codon stop., Si un EJC est trouvé plus de 50 nucléotides après (ou « en aval ») d’un codon stop, la cellule reconnaît que cela est aberrant et détruit l’ARNm (Popp et Maquat, 2013; Figure 1B).
la région d’ARNm en aval du codon stop est connue sous le nom de Région 3′ non traduite (3’UTR). Bien que cette région ne Code aucune protéine, elle est importante pour réguler l’activité de la molécule d’ARNm. Les microARN sont de courtes molécules D’ARN non codantes (c’est-à-dire qu’elles ne codent pas pour la protéine) qui se lient aux sites 3’UTR d’une manière spécifique à la séquence., La liaison des microARN peut inhiber la traduction et encourager la dégradation d’une molécule d’ARNm, et est très importante pour contrôler comment et quand les gènes sont exprimés chez les eucaryotes (Fabian et Sonenberg, 2012).
Zhao et coll. raisonné que si un ARNm contient une mutation non-sens, la région codante en aval de la mutation sera essentiellement convertie en un 3’UTR (Zhao et al., 2014). Ce 3’UTR étendu pourrait contenir des sites de liaison pour les microARNs, qui se lieraient à et réguleraient la molécule d’ARN messager. Zhao et coll., a utilisé une série d’expériences élégantes pour confirmer cette hypothèse et, ce faisant, a identifié une nouvelle méthode de désintégration non-sens qui est dirigée par les microARN (Figure 1C). Tout d’abord, Zhao et coll. insertion d’un site de liaison de microARN dans la région codante d’un ARNm. Cela n’a eu aucun effet sur l’ARNm, confirmant l’opinion dominante selon laquelle la régulation des microARN ne se produit pas efficacement dans les régions codantes (Gu et al., 2009). Cependant, lorsque Zhao et al. introduit un codon non-sens de sorte que le site de microARN était maintenant situé dans le 3’UTR, microARN pourrait se lier à lui, ce qui a induit la désintégration de l’ARNm (Zhao et al., 2014).,
Zhao et coll. nous avons ensuite montré que les microARN influencent également la désintégration Non-sensée des ARNm naturels. Ils se sont concentrés sur L’APC, un gène suppresseur de tumeur qui contient souvent des mutations absurdes dans les tumeurs du cancer colorectal. Le gène APC a été un choix particulièrement judicieux pour cette étude, car la plupart de ses mutations non-sens se produisent dans un point chaud du dernier exon, de sorte qu’elles ne sont pas reconnues par la méthode complexe de la jonction exon de la désintégration Non-sensée (Miyoshi et al., 1992). Cependant, Zhao et coll., trouvé que les mutations non-sens dans cette région exposent les sites de liaison des microARN dans le 3’UTR nouvellement formé, ce qui conduit à la désintégration de l’ARNm copié à partir du gène APC. Enfin, Zhao et coll. on a utilisé des gènes rapporteurs basés sur le gène suppresseur de tumeur associé au cancer du sein BRCA1, qui est sensible à la forme EJC de la désintégration Non-sensée, pour montrer que le complexe de jonction exon et les mécanismes microARN de la désintégration Non-sensée peuvent tous deux fonctionner sur le même ARNm (Zhao et al., 2014).
Les conclusions de Zhao et al., révélez une nouvelle fonction intrigante pour les microARN et un nouveau mécanisme de désintégration Non-sensée. Ils soulèvent également plusieurs questions importantes. Quelle est la contribution relative de la désintégration induite par le non – sens de L’EJC et du microARN dans les cellules? Contrairement à la désintégration médiée par le non-sens EJC, la régulation des microARN a tendance à être assez faible (Leung et Sharp, 2010). Cependant, plusieurs sites de microARN fonctionnent de manière coopérative, de sorte que le niveau de désintégration médiée par le non-sens des microARN est susceptible d’être fortement influencé par le nombre et l’identité des sites de microARN révélés par une mutation non-sens., En outre, l’expression des microARN est fortement régulée pendant le développement et dans certains tissus, ainsi que dans les cancers, ce qui suggère que la désintégration non-sens-médiée par les microARN est également susceptible d’être étroitement régulée.
D’autres méthodes de désintégration non-sens indépendante de L’EJC ont également été découvertes (Metze et al., 2013), alors comment la méthode des microARN interagit-elle avec ces mécanismes alternatifs? En régulant les protéines impliquées dans la désintégration non-sens du complexe de jonction d’exons, les microARN peuvent influencer le déroulement de la désintégration (Bruno et al., 2011)., De plus, une nouvelle étude a montré que la liaison des microARN peut créer une cible appropriée pour la désintégration Non-sensée de L’EJC en modifiant la façon dont le ribosome lit une molécule d’ARNm spécifique (CCR5, qui code une protéine utilisée par de nombreuses formes de VIH pour envahir les cellules; Belew et al., 2014). Dans l’ensemble, il semble que la complexité de la façon dont la cellule traite les mutations absurdes et comment les microARN sont impliqués commence seulement à être révélée.